Notícias
RedeVírus-MCTI realiza avaliação das variantes de SARS-COV 2 circulantes no Estado de SP

INFORME N. 25 REDE CORONA-ÔMICA.BR-MCTI
A Rede Corona-ÔmicaBR-MCTI, por meio do Laboratório de Pesquisa em Virologia da Faculdade de Medicina de São José do Rio Preto (FAMERP), juntamente com o Instituto de Biotecnologia (IBTEC) da UNESP Botucatu; o Instituto de Biociências, Letras e Ciências Exatas (IBILCE) da UNESP, São José do Rio Preto; e a Faculdade de Zootecnia e Engenharia de Alimentos da USP Pirassununga (FZEA-USP), reporta os resultados do sequenciamento de amostras positivas para SARS-CoV-2 de pacientes provenientes de cidades pertencentes ao Departamento Regional de Saúde de São José do Rio Preto – DRS XV (Figura 1a).
Desde o início do projeto de vigilância genômica, foram gerados 574 genomas completos de SARS-CoV-2, provenientes de 26 cidades da região. A amostragem compreendendo o período de outubro de 2020 a junho de 2021 (390 genomas) detectou 14 variantes circulantes em São José do Rio Preto e região, as quais foram classificadas em B.1 (n=1, 0,25%), B.1.1 (n=3, 0,77%); B.1.1.273 (n=1, 0,25%), B.1.1.28 (n=27, 7%), B.1.1.33 (n=5, 1,3%), B.1.1.332 (n=1, 0,25%), B.1.1.393 (n=1, 0,25%), B.1.2 (n=1, 0,25%), B.1.1.7 (n=4, 1%), N.9 (n=13, 3,3%), P.1 (n=291, 74,6%), P.1.1 (n=2, 0,5%), P.1.7 (n=3, 0,77%) e P.2 (n=37, 9,5%).
Em uma análise mais recente, realizada de forma randomizada, no período correspondente 01 de julho a 11 de agosto de 2021 e que incluiu 16 cidades da DRS XV (Bady Bassitt, Bálsamo, Catanduva, Cedral, Guapiaçu, Macaubal, Mirassol, Neves Paulista, Nova Aliança, Palestina, Tanabi, Uchoa, Urupês, Valentim Gentil, Votuporanga e São José do Rio Preto), foram gerados 184 genomas completos, os quais foram classificados em seis linhagens pelo software Pangolin COVID-19 Lineage Assigner Tool (Figura 1b). A variante P.1 (Gamma) apresentou maior prevalência entre os genomas sequenciados (n= 148, 80,4%), seguida da linhagem P.1.7 (n= 29, 15,8%), a qual é descendente de P.1 (Figura 1b). No mês de julho, em menor frequência, foram detectadas a variante de preocupação (VOC) B.1.1.7 (Alpha) (n =1, 0.54%) e a variante P.4 (n =1, 0,54%), a qual emergiu recentemente no interior do estado de São Paulo e possui a mutação L452R na proteína S, que tem grande impacto biológico e está presente na variante de preocupação Delta. Em amostras do mês de agosto, além das variantes P.1 e P.1.7, foram identificadas a linhagem P.1.2 (n= 1, 0,54%) e quatro genomas classificados como B.1.617.2 (2,1%).
Dentre os achados mais relevantes, pode-se destacar a introdução e primeira detecção da VOC Delta B.1.617.2, em São José do Rio Preto. Além disso, é notável a dominância da variante P.1 no município de São José do Rio Preto e nas demais cidades pertencentes a DRS XV, bem como de sua linhagem descendente P.1.7, a qual foi detectada pela primeira vez na região em junho de 2021 e apresentou um aumento em frequência a partir de julho de 2021. Esses dados ressaltam a importância da constante vigilância das variantes circulantes, bem como o monitoramento da prevalência da variante Delta recentemente introduzida na região. Dessa forma, medidas eficazes para o controle e enfrentamento da pandemia podem ser tomadas, a fim de prevenir a disseminação do vírus e um aumento em número de casos.
Todas os genomas completos gerados encontram-se disponíveis na base de dados EpiCoV
em GISAID (https://www.gisaid.org).
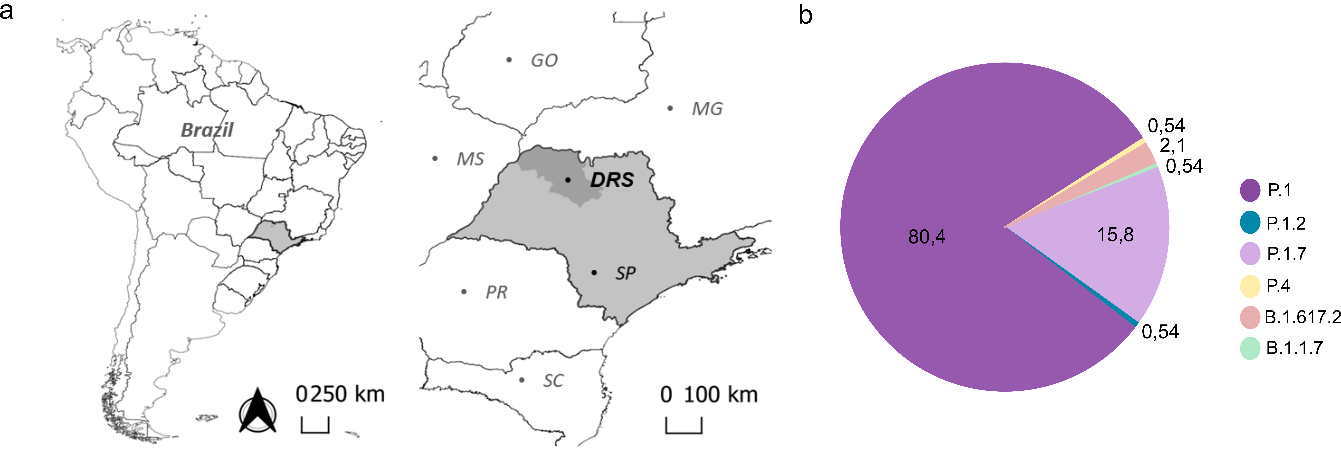
Figura 1. Vigilância genômica de SARS-CoV-2 realizada em cidades pertencentes ao Departamento Regional de Saúde de São José do Rio Preto – DRS XV. a) Mapa geopolítico do Brasil, com destaque para o estado de São Paulo. Em maior escala, o mapa do estado de São Paulo em cinza claro, com a área correspondente à DRS XV em destaque (cinza escuro). b) Classificação e prevalência das linhagens circulantes na DRS XV entre os meses de julho e agosto de 2021
Rede Corona-Ômica.BR-MCTI