Notícias
Corona-ômica BR MCTI estuda a dispersão do vírus SARS-CoV-2 no estado do Acre e identifica introdução da linhagem VOI lambda C.37.

A Rede Vírus MCTI informa que o Laboratório de Bioinformática (LABINFO) do Laboratório Nacional de Computação Científica (Petrópolis-RJ), um instituto de pesquisa do Ministério de Ciência e Tecnologia (MCTI), em colaboração com Centro de Infectologia Charles Mérieux/Laboratório Rodolphe Mérieux, FUNDHACRE e o Laboratório Central de Saúde Pública - LACEN do Estado do Acre vem informar que foi realizado sequenciamento genético de 95 amostras coletadas aleatoriamente do vírus SARS-CoV-2. As amostras foram coletadas entre 16 de Março e 29 de Julho no Estado do Acre dos seguintes municípios: Acrelândia (5), Assis Brasil (12), Brasileia (10), Capixaba (5), Epitaciolândia (5), Feijó (5), Plácido de Castro (3), Porto Acre (2), Rio Branco (37), Sena Madureira (2), Tarauacá (4), Xapuri (5).
Oitenta e duas amostras foram identificadas sendo a VOC Gamma P.1 (86,32%), oito amostras da VOI Lambda C.37 (8,42%) duas da P.1.2 (2,11%) e uma da amostra da P.1.1, P.1.4, B.57 (1,05%) respectivamente (Figura 1).
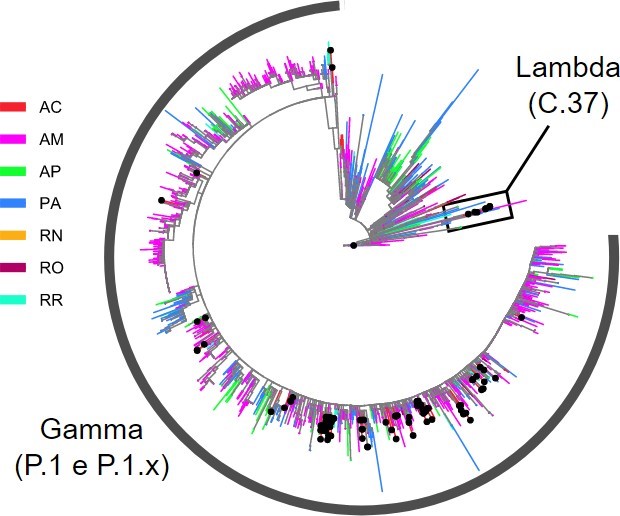
Figura 1: Árvore evolutiva de todas as sequências disponíveis e de boa qualidade provindas da região Norte do país, disponíveis na base de dados GISAID no dia 23/08/2021. Em destaque, as variantes Gamma e Lambda.
Ao serem comparadas com sequências da região Norte do Brasil, podemos observar 3 grupamentos de sequências de P.1 no estado (Figura 2). Devido a grande dispersão da linhagem P.1 por estes territórios, não é possível precisar a origem destas introduções.
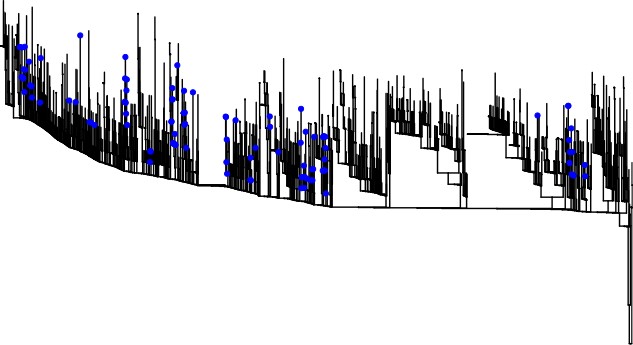
Figura 2: Árvore evolutiva de todas as sequências da variante P.1 (Gamma) disponíveis e de boa qualidade provindas da região Norte do país, disponíveis na base de dados GISAID no dia 23/08/2021. Em azul, sequências provenientes do estado do Acre, demonstrando ao menos três conjuntos diferenciados.
Foi ainda encontrado oito amostras da linhagem VOI Lambda C.37 que ainda não tinha sido identificada na região norte do país. Essa linhagem tem somente 7 genomas identificados no Brasil, sendo duas no Rio Grande do Sul e cinco no Estado de São Paulo. Análises filogenéticas preliminares mostram que as amostras brasileiras pertencem a dois clados diferentes, indicando que ocorreram ao menos dois eventos de introdução no Acre (Figura 3). Uma dessas introduções pode estar relacionada a sequências de São Paulo, mas análises mais profundas são necessárias para confirmar ou não essa relação. nenhuma das sequências do Acre está relacionada a sequências do Rio Grande do Sul.
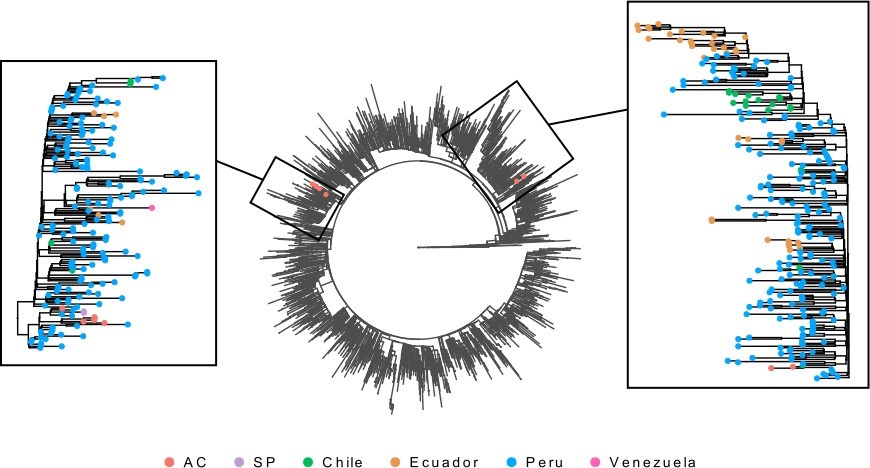
Figura 3. Árvore filogenética de todas as amostras da linhagem C.37 (Lambda) com boa qualidade disponíveis na base de dados GISAID. É possível observar que ocorreram duas introduções da variante no Acre.
Devido ao fato dessa no VOI Lambda do SARS-CoV-2 estar circulando no Estado do Acre, fazemos este comunicado ao mesmo tempo que as análises ainda estão sendo finalizadas. Em breve os genomas serão depositados em bases de dados públicas e o trabalho será submetido em periódico internacional.
A coordenadora do Laboratório de Bioinformática do LNCC, Ana Tereza Vasconcelos, chama a atenção que a cooperação institucional possibilitou realizar a vigilância epidemiológica genômica de grande amplitude geográfica no Estado do Acre e que esta iniciativa quadruplicou o número de genomas sequenciados até o momento. Alerta ainda, que estudos sobre a dispersão do Sars-Cov-2 são extremamente importantes para a segurança de todos.
Rede Corona-Ômica.BR-MCTI