Notícias
REDEVÍRUSMCTI
Rede Corona-Ômica.BR-MCTI realiza trabalho de mapeamento de variantes de SARS-CoV-2 circulantes em 12 cidades do estado do Tocantins

A Rede Vírus MCTI informa que a Rede Corona-Ômica.BR-MCTI, através do Laboratório de Bioinformática & Biotecnologia (Labinftec) da Universidade Federal do Tocantins (UFT) em colaboração com o Laboratório Central de Saúde Pública do Tocantins (LACEN-TO) e Laboratório de Baculovírus da Universidade de Brasília (UNB) identificou e sequenciou 24 genomas de SARS-CoV-2 de amostras coletadas em 12 cidades do estado do Tocantins (Tabela 1).
Informamos que, de 24 amostras sequenciadas nessa região, 18 (75% das amostras sequenciadas)foram classificadas como P.1 pelo Pango Lineages e seis (25%) como P.1.2 (Tabela 1 e Figura 1). A variante do vírus SARS-CoV-2 denominada P.1 teve origem na região amazônica e está associada a uma maior transmissibilidade quando comparado as variantes que prevaleciam em circulação no país (Naveca et al., 2021; Faria et al., 2021). A variante P.1 tem se espalhado rapidamente por muitos estados brasileiros, demonstrando predomínio dentre as variantes de preocupação (VOCs – Variants of Concern ) em circulação no país (para mais detalhes veja 4º Relatório Técnico de Situação – Projeto Corona-Ômica-BR – 06/05/2021). Isso reforça a grande importância de subsídios e recursos para vigilância epidemiológica e genômica para rastreamento rápido da disseminação de VOCs, com fornecimento de respostas rápidas para os sistemas de saúde pública. Recentemente, novas mutações foram observadas na variante P.1, dando origem a uma nova sub-linhagem P.1.2 (Almeida et al., 2021). Essa variante P.1.2 ou P1-like II foi identificada inicialmente no RJ e abriga novas mutações em relação a P1. Ela está se espalhando em nove estados de quatro regiões brasileiras (Norte = 2, Nordeste = 1, Sul = 35 e Sudeste = 13) e abriga 15 mutações definidoras de linhagem P.1 e sete mutações únicas ( https://github.com/cov-lineages/pango-designation/issues/77 ).
Ressaltamos também que identificamos nesses genomas 42 mutações do tipo SNPs (Polimorfismos de Nucleotídeo Único) e duas mutações do tipo InDel (Inserção-Deleção). Dentre os 42 SNPs identificados, 41 são em regiões codificantes do genoma viral e um na porção 5’UTR (C241T). Onze mutações na região codificante foram caracterizadas como sinônimas ou silenciosas e 30 destas foram preditas com substituições de aminoácidos. Dentre essas 30 substituições, 43,3% são localizadas no gene S (sendo 13 mutações pontuais: L18F, T20N, P26S, D138Y, R190S, K417T, E484K, N501Y, D614G, H655Y, P681H, T1027I, V1176F). A ORF1ab que compreende aproximadamente 67% do genoma e codifica 16 proteínas não-estruturais apresentou um total de 8 mutações com substituições de aminoácidos (S1188L, K1230N, K1795Q, S1924I, T2846I, P4715L, E5665D, M6661I). Seis substituições são localizadas no gene N (P80R, T135I, S202C, S202T, R203K, G204R). Três foram encontradas na ORF3a (L106F) e um na ORF8 (E92K).
Desta forma, recomendamos que as providências cabíveis sejam tomadas pelos órgãos estaduais e federais competentes e agradecemos a colaboração com o Laboratório Central de Saúde Pública do Tocantins (LACEN-TO) e Laboratório de Baculovírus da Universidade de Brasília (UNB) na obtenção dos presentes resultados.
Tabela 1. Localidade, data de coleta e variante identificada nas 24 amostras provenientes de 12 cidades do estado do Tocantins.
|
Remessa – Lacen-TO |
Cidade |
N de amostras |
Data de coleta |
Pango lineage |
|
6/1604 |
Formoso do Araguaia |
1 |
26/05/2021 |
P.1 |
|
9/1614 |
Palmas |
1 |
27/05/2021 |
P.1 |
|
25/1614 |
Palmas |
1 |
27/05/2021 |
P.1 |
|
27/1614 |
Palmas |
1 |
27/05/2021 |
P.1.2 |
|
42/1614 |
Porto Nacional |
1 |
26/05/2021 |
P.1.2 |
|
69/1614 |
Palmas |
1 |
27/05/2021 |
P.1 |
|
72/1614 |
Palmas |
1 |
27/05/2021 |
P.1 |
|
94/1614 |
Palmas |
1 |
27/05/2021 |
P.1 |
|
39/1615 |
Porto Nacional |
1 |
26/05/2021 |
P.1 |
|
57/1615 |
Porto Nacional |
1 |
27/05/2021 |
P.1 |
|
10/1616 |
Jau do Tocantins |
1 |
27/05/2021 |
P.1 |
|
60/1616 |
Gurupi |
1 |
27/05/2021 |
P.1 |
|
36/1617 |
Gurupi/Residência Em Goiânia |
1 |
26/05/2021 |
P.1.2 |
|
91/1617 |
Lagoa da Confusão |
1 |
27/05/2021 |
P.1 |
|
71/1618 |
Rio dos Bois |
1 |
27/05/2021 |
P.1.2 |
|
88/1618 |
Fortaleza do Tabocão |
1 |
27/05/2021 |
P.1.2 |
|
41/1671 |
Novo Alegre |
1 |
31/05/2021 |
P.1 |
|
73/1671 |
Palmas |
1 |
01/06/2021 |
P.1 |
|
10/1672 |
Palmas |
1 |
01/06/2021 |
P.1 |
|
29/1672 |
Palmas |
1 |
01/06/2021 |
P.1 |
|
68/1672 |
Paraiso do Tocantins |
1 |
31/05/2021 |
P.1.2 |
|
5/1674 |
Palmas |
1 |
01/06/2021 |
P.1 |
|
77/1674 |
Rio Sono |
1 |
31/05/2021 |
P.1 |
|
79/1675 |
Brejinho de Nazaré |
1 |
01/06/2021 |
P.1 |
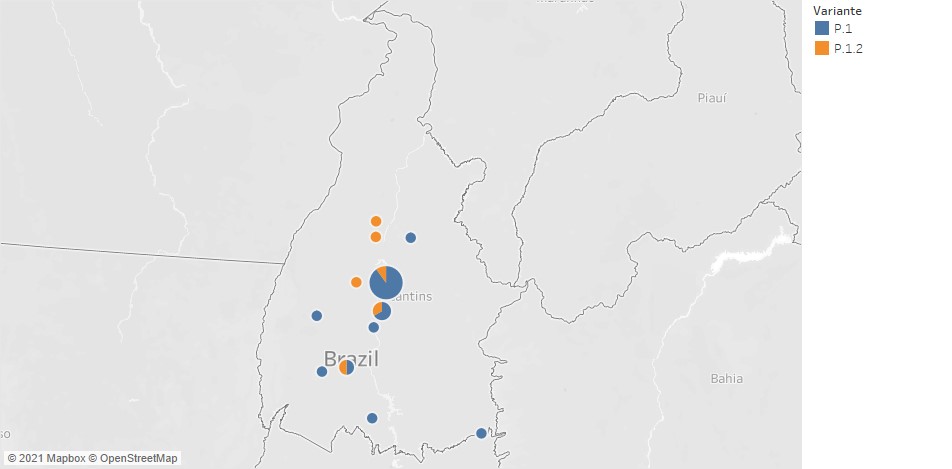
Figura 1. Mapa baseado em Longitude e Latitude. A cor mostra detalhes sobre as variantes P.1 e P.1.2 em circulação no estado. O tamanho mostra soma de N.
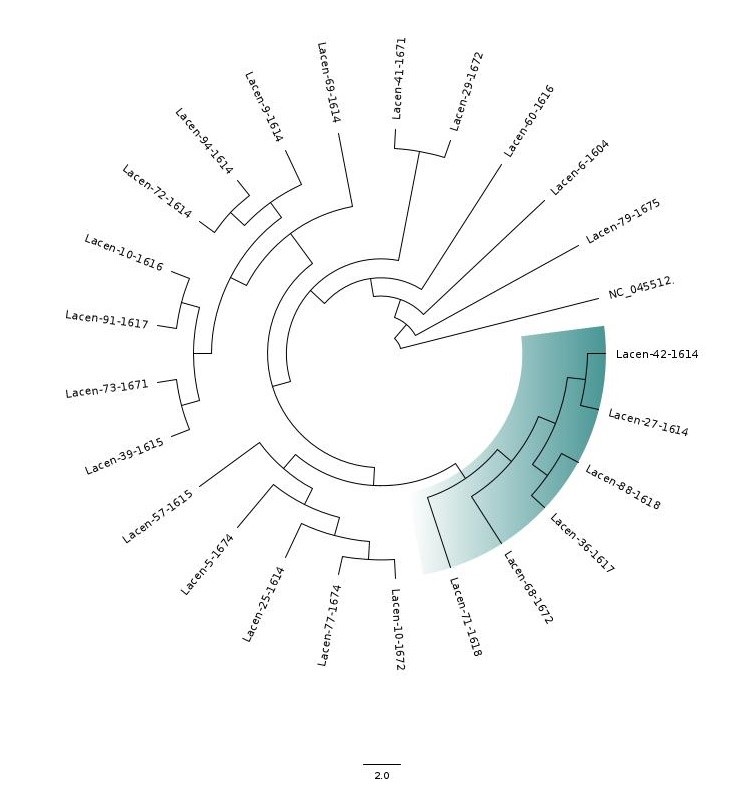
Figura 2. Árvore filogenética de Maximum Likelihood reconstruída para as 24 amostras sequenciadas e genoma referência (NC_045512). Em azul destacamos as seis amostras da linhagem P.1.2.
A EQUIPE
Universidade Federal do Tocantins (UFT)
Fabrício Souza Campos
Ueric José Borges de Souza
Raíssa Nunes dos Santos
Universidade de Brasília (UNB)
Bergmann Morais Ribeiro
Fernando Lucas Melo
Aline Belmok de Araújo Dias Locca
Laboratório Central de Saúde Pública do Tocantins (LACEN-TO)
Jucimária Dantas Galvão
Sirlene Borges Damasceno
Tereza Cristina Vieira de Rezende
REFERÊNCIAS
Almeida LGP, Lamarca AP, et al. Genomic Surveillance of SARS-CoV-2 in the State of Rio de Janeiro, Brazil: a technical briefing. nCoV-2019 Genomic Epidemiology. 2021. Disponível em: https://virological.org/t/genomic-surveillance-of-sars-cov-2-in-the-state-of-rio-de-janeiro-brazil-technical-briefing/683
Naveca F, Nascimento V, Souza V, Corado A,Nascimento F, Silva G, et al. COVID-19 epidemic in the Brazilian state of Amazonas was driven by longterm persistence of endemic SARS-CoV-2 lineages and the recent emergence of the new Variant of Concern P.1. 2021. https://doi.org/10.21203/rs.3.rs-275494/v1
Faria NR, Mellan TA, Whittaker C, Claro IM, Candido DS, Mishra S, et al. Genomics and epidemiology of a novel SARS-CoV-2 lineage in Manaus, Brazil. medRxiv. 2021. https://doi.org/10.1101/2021.02.26.21252554
RedeCorona-Ômica.BR-MCTI