Notícias
REDEVÍRUS MCTI
Rede Corona-ômica BR MCTI estuda a dispersão do vírus SARS-CoV-2 no estado do Acre e identifica linhagem ainda não descrita no Brasil

A Rede Vírus MCTI informa que a Rede Corona-Ômica.BR-MCTI, através do Laboratório de Bioinformática (LABINFO) do Laboratório Nacional de Computação Científica (Petrópolis-RJ), um instituto de pesquisa do Ministério de Ciência e Tecnologia (MCTI), em colaboração com Centro de Infectologia Charles Mérieux/Laboratório Rodolphe Mérieux, FUNDHACRE, realizou o sequenciamento genético de 95 amostras do vírus SARS-CoV-2 e identificou linhagem ainda não descrita no Brasil. As amostras foram coletadas entre 05 de janeiro e 12 de abril no Estado do Acre.
Segundo os dados coletados, a maior parte das amostras são da linhagem P1 (63, Figura 1). Ao serem comparadas com sequências da região Norte do Brasil e países vizinhos, podemos observar ao menos 6 introduções no estado (Figura 2). Devido a grande dispersão da linhagem P.1 por estes territórios, não é possível precisar a origem destas introduções.
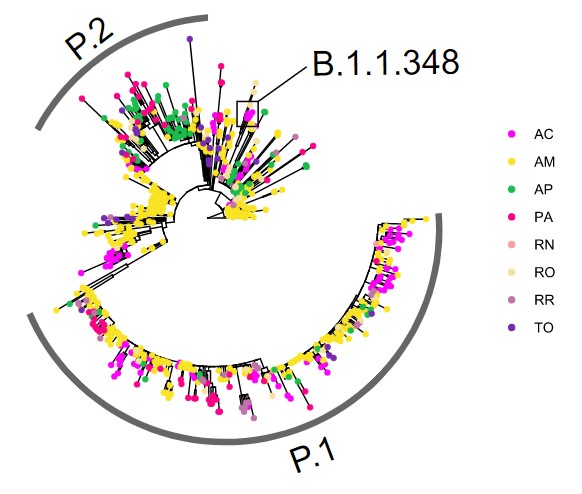
Figura 1: Árvore evolutiva de todas as sequências disponíveis e de boa qualidade provindas da região Norte do país, amostradas entre 05/01/2020 e 20/04/2021. Em destaque, as linhagens P.1, P.2 e B.1.1.348.
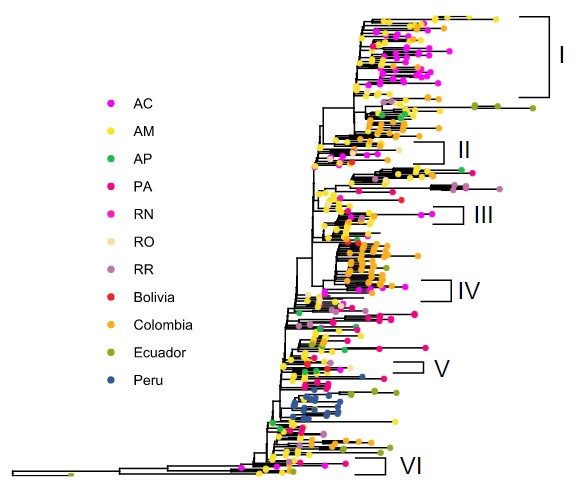
Figura 2: Árvore evolutiva da linhagem P.1 na região Norte do Brasil e países vizinhos, construída com sequências disponíveis e de boa qualidade amostradas entre 04/12/2020 e 12/05/2021. Os números apontam diversos conjuntos de sequências do Acre distribuídos ao longo da filogenia sugerindo que ocorreram diversos eventos de introdução da linhagem no estado.
No estudo também foram identificadas quatro amostras da linhagem B.1.1.348 que ainda não tinha sido identificada em território brasileiro. Essa linhagem já havia sido identificada nos EUA e, a partir de dezembro de 2020, foi encontrada em vários países da América do Sul. Análises filogenéticas preliminares mostram que as amostras brasileiras formam um único clado, indicando que houve apenas um evento de introdução no país.
Dentre todas as sequências desta linhagem disponíveis e com qualidade (n = 792), as sequências brasileiras são evolutivamente mais relacionadas a uma amostra da cidade de Lima no Peru, país que faz fronteira com o estado do Acre. Isto indica que o país é a possível origem das sequências brasileiras da linhagem B.1.1.348. No entanto, é importante ressaltar que esta interpretação é altamente dependente da amostragem em outros países.
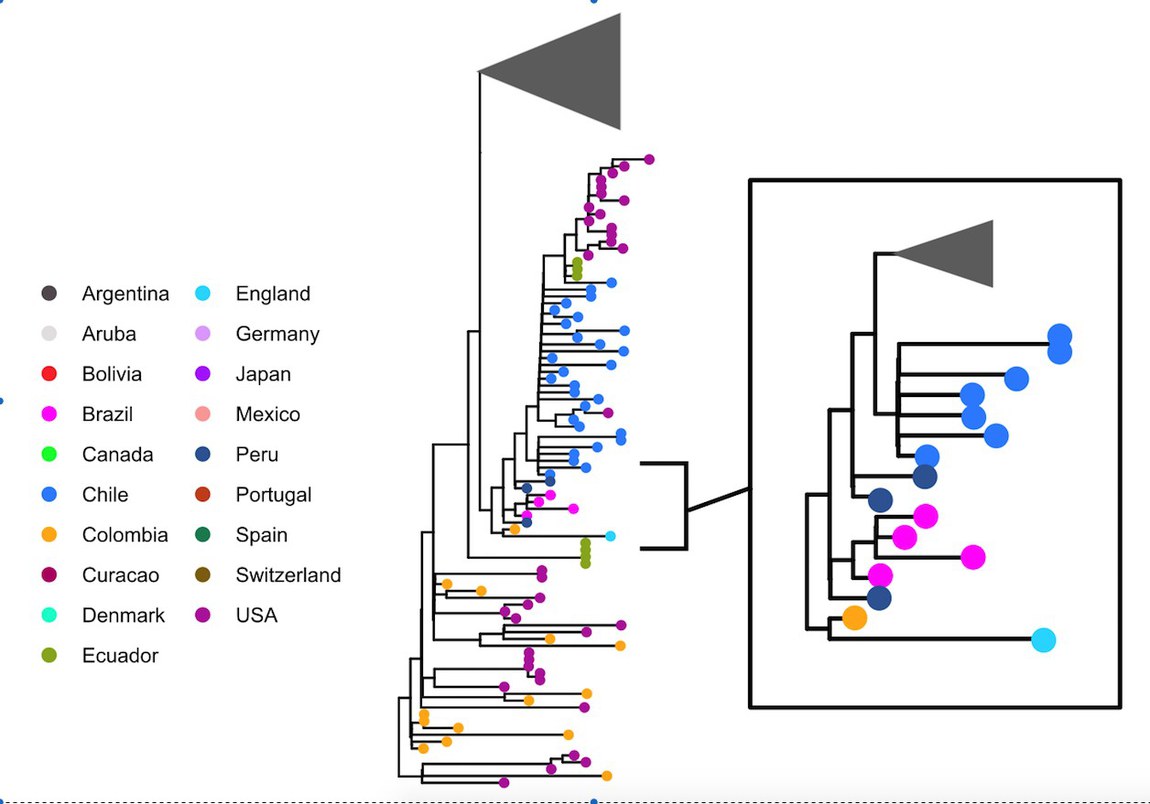
Figura 3: Árvore evolutiva da linhagem B.1.1.348, demonstrando a relação entre sequências do Peru (azul) e do Brasil (rosa). A reconstrução utiliza apenas sequências de boa qualidade amostradas entre 30/04/2020 e 13/05/2021 Os triângulos representam sequências removidas da figura para facilitar a visualização.
Dentre as mutações que definem esta linhagem foi identificada uma mutação na proteína Spike G1167A que havia sido encontrada na linhagem B.1.177 após a primeira onda da epidemia na Espanha. Estudos ainda não publicados sugerem que essa mutação não afeta a estabilidade térmica da proteína ou a suscetibilidade de anticorpos em indivíduos vacinados.
Devido ao fato dessa nova linhagem do SARS-CoV-2 não ter sido ainda identificada no Brasil, fazemos este comunicado ao mesmo tempo que as análises ainda estão sendo finalizadas. Em breve os genomas serão depositados em bases de dados públicas e o trabalho será submetido em periódico internacional.
Ainda de acordo com os pesquisadores do LNCC, os dados gerados neste trabalho da Rede Corona-ômica-MCTI, triplicaram o número de amostras depositadas em bases públicas do estado do Acre (ref. GISAID 01/06/2021). Alerta ainda, que estudos sobre a dispersão do Sars-Cov-2 são extremamente importantes para a segurança de todos.
Rede Corona-Ômica.BR-MCTI